研究人员使用深度学习将2D图像转换为3D
加州大学洛杉矶分校的研究小组设计了一种技术,可以扩展荧光显微镜的功能,该技术使科学家能够使用在特殊照明下发光的染料精确标记活细胞和组织的一部分。研究人员利用人工智能将二维图像转换成虚拟三维切片的堆栈,这些三维切片显示了生物体内的活动。
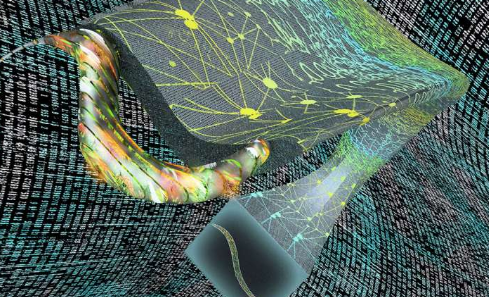
在《自然方法》上发表的一项研究中,科学家们还报告说,他们的名为“ Deep-Z”的框架能够修复图像中的错误或像差,例如当样品倾斜或弯曲时。此外,他们证明了该系统可以从一种类型的显微镜拍摄2D图像并虚拟地创建样品的3D图像,就好像它们是由另一台更高级的显微镜获得的一样。
加州大学洛杉矶分校(UCLA)校长电气教授Aydogan Ozcan说:“这是一种非常强大的新方法,通过深度学习可以对活体标本进行3D成像,同时使对样品的毒性最小的光暴露最少。”加州大学洛杉矶分校加州大学纳米系统研究所的计算机工程和副主任。
除了使标本免于潜在的有害剂量照射外,该系统还可以为生物学家和生命科学研究人员提供一种新的3-D成像工具,该工具比当前方法更简单,更快且更便宜。校正像差的机会可能使研究活生物体的科学家能够从图像中收集数据,否则这些图像将无法使用。研究人员还可以虚拟访问昂贵且复杂的设备。
这项研究基于Ozcan及其同事开发的较早技术,该技术使他们能够以超分辨率渲染二维荧光显微镜图像。两种技术都依靠深度学习来提高显微技术的准确性,即使用数据来“训练”神经网络,即受人脑启发的计算机系统。
Deep-Z是使用来自扫描荧光显微镜的实验图像教授的,该图像可以在多个深度聚焦,以实现样品的3D成像。在数千次训练中,神经网络学习了如何拍摄2D图像并推断出样品中不同深度的准确3D切片。然后,对框架进行了盲目测试-注入了不是其训练内容的图像,并且将虚拟图像与从扫描显微镜获得的实际3-D切片进行了比较,从而提供了出色的匹配度。
Ozcan和他的同事将Deep-Z应用于秀丽隐杆线虫的图像,秀丽隐杆线虫由于其简单易懂的神经系统而成为神经科学中的常见模型。研究人员将蠕虫的2D电影逐帧转换为3D电影,从而能够跟踪蠕虫体内单个神经元的活动。然后从在不同深度拍摄的秀丽隐杆线虫的一幅或两幅2-D图像开始,Deep-Z生成虚拟的3-D图像,使团队可以识别蠕虫中的单个神经元,与扫描显微镜的3-D输出相匹配,除了与生物接触的光更少。
研究人员还发现,即使仅使用与样品表面完全平行的3-D切片训练神经网络,Deep-Z仍可以从样品倾斜或弯曲的2-D表面生成3-D图像。
UCLA研究生,该出版物的第一作者,作者Yichen Wu表示:“这一功能实际上非常令人惊讶。” “有了它,您可以看透曲率或其他复杂的成像难题。”
在其他实验中,使用来自两种荧光显微镜的图像对Deep-Z进行了训练:宽视场,它将整个样本暴露在光源下;共聚焦,它使用激光逐部分扫描样品。Ozcan和他的团队表明,他们的框架可以使用样品的2D宽视场显微镜图像生成与共聚焦显微镜拍摄的图像几乎相同的3D图像。
这种转换很有价值,因为与宽视野相比,共聚焦显微镜可以产生更清晰,对比度更高的图像。另一方面,宽视野显微镜以较少的费用和较少的技术要求捕获图像。
加州大学洛杉矶分校(UCLA)电气与计算机工程学助理教授兼第一作者Yair Rivenson说:“这个平台通常适用于各种成对的显微镜,而不仅仅是宽视野到共焦的转换。” “每种显微镜都有其优点和缺点。有了这个框架,您可以使用AI以数字方式连接不同类型的显微镜,从而实现两全其美。”
其他作者是加州大学纳米系统研究所高级光显微学和光谱实验室科学总监Eyal Ben-David和Laurent Bentolila的博士后研究生,Hongda Wang和Yilin Luo。以色列希伯来大学的克里斯蒂安·普里茨(Christian Pritz)。
推荐内容
-
面部注氧有什么作用
-2022年4月28日发(作者:鼻子两侧红血丝)面部注氧有什么作用关于《面部注氧有什么作用》,是我们特意为大家整理的,希望对大家有所帮助。脸
-
12月2日广西疫情最新数据公布 广西昨日新增境外输入确诊8例
疫情期间,请广大市民朋友们密切关注国内中、高风险地区信息动态,如非必要,请勿前往疫情发生地区,广西卫健委网站通报,12月1日0至24时,
-
思连康\思密达联合治疗新生儿母性黄疸
-2022年4月18日发(作者:食物中毒症状)思连康\思密达联合治疗新生儿母性黄疸作者单位:455133河南省安阳县第一人民医院通讯作者:申琴玉目
-
12月8日浙江内蒙古黑龙江上海疫情最新数据 31省昨日新增本土病例44例
现在国内疫情具体情况如何?根据国家卫健委公布数据得知,在过去的一天时间里31省新增本土病例38+5例的消息引起了广泛网友的关注,具体是什么
-
肚脐眼周围疼痛的原因
-2022年4月16日发(作者:肺癌的治疗)如对您有帮助,可购买打赏,谢谢肚脐眼周围疼痛的原因导语:肚脐周围出现不正常的症状是一定要引起我们
-
Synbiosis将在ASM Microbe 2019上展示新的AutoCOL全自动菌落计数系统
Synbiosis是一家历史悠久的专业自动微生物系统制造商,很高兴地宣布其新的AutoCOL全自动菌落计数系统将于6月21日至23日在ASM Microbe 201
-
U研究开始揭开hagfish的僵尸心中的神秘面纱
Hagfish是粘糊糊的底栖居民,靠死海生物的尸体生活,并在难以获得氧气的深水中茁壮成长。事实上,他们的心脏可以在没有任何氧气的情况下继
-
9月6日扬州疫情最新实时消息公布 截至目前扬州537例患者已治愈出院
【摘要】非必要不外出、不出行、不远行。如果您28天内有境外或14天内有国内中高风险区的旅居史,请及时向社区报告。无锡集结完毕,准备出发
-
2021QS世界大学排名 清华位列亚洲排名第三
昨日一则关于2021QS世界大学排名的相关消息引起了网上热议,想必大家也是比较好奇了,下面小编就带大家一起去了解一下事情始末吧?10日,QS
-
8月16日澳门疫情最新实时消息公布 澳门18日零时起解除关闭部分娱
澳门(葡语Macau、英语Macao),简称澳,全称中华人民共和国澳门特别行政区,位于中国南部珠江口西侧,是中国大陆与南中国海的水陆交汇处,毗




