新的tRNA测序方法显示微生物组活性
一种新的先进的、高通量的测序技术应该使科学家能够更牢固地掌握自然产生的微生物在现实环境和饮食中的活性。芝加哥大学的研究人员开发了直接分析转移RNA (tRNA)的新技术,为微生物群落对各种环境变化的反应提供了一个更清晰的图像,这些环境变化包括温度变化或营养物质有效性的变化。这项新研究的发现发表在今天的《自然通讯》杂志上,文章题为《高通量转移RNA测序和修饰分析的微生物组特征》。
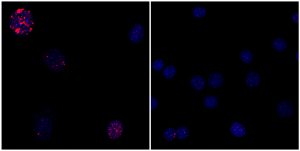
在目前的研究中,研究小组展示了tRNA测序在低脂或高脂饮食小鼠肠道微生物组样本中的应用。利用这项研究中描述的新软件和计算策略,创建了从肠道样本中提取的tRNA分子的目录,追踪它们到负责其表达的细菌,并测量转录后tRNA中发生的化学修饰。
作者写道:“我们报告了一种直接测序方法,tRNA-seq,以及一套软件,tRNA-seq工具,用于恢复微生物tRNA转录本的序列、丰度谱和转录后修饰。”“我们使用tRNA-seq对cecal样本进行分析,以类似于16S核糖体RNA基因扩增子的方式区分高脂和低脂喂养的小鼠,并揭示tRNA修饰中的分类单元和饮食依赖变异。”我们的研究结果为复杂环境微生物体内tRNA基因表达和转录后修饰的动力学提供了特异性的原位观察。
细菌中的每个tRNA平均有8种化学修饰,可以调节其功能。新的高通量测序和分析策略可以检测到其中的两种,但它也可以在每个位点测量从0到100%的修改量。其中一种被称为m1A的修饰,在喂食高脂肪食物的小鼠肠道微生物组中含量更高。这是科学家第一次能够在任何微生物组中看到tRNA的修饰水平的变化。
芝加哥大学生物化学和分子生物学教授、高级研究员潘涛博士指出:“我们的研究是倒退的。”“我们对m1A tRNA的修饰为何存在以及它们的作用没有先期的概念,但在微生物组中看到任何修饰的改变都是前所未有的。”
m1A的修饰有助于合成某些类型的蛋白质,这些蛋白质在高脂肪饮食中可能更为丰富。研究人员还不知道这些改变是否会对饮食产生反应,或者它们是否已经存在并变得活跃,从而促进这些蛋白质的合成。
这项研究是芝加哥大学由凯克基金会资助的一系列微生物组项目中的第一个。潘博士是使用tRNA测序工具的先驱,这笔赠款将资助继续开展的工作,通过芝加哥大学(University of Chicago)医学助理教授、联合高级研究研究员a·穆拉特·埃伦(A. Murat Eren)博士开发的新计算策略,让tRNA测序工具能够被广泛使用。tRNA测序产生的大量数据可以以低成本提供与人类或环境相关的微生物的关键见解。
“过去20年里出现的分子和计算方面的进步,只帮助我们揭开了微生物生命及其对周围环境的影响的表面,”埃伦博士说。”通过提供快速和负担得起的见解转化的核心机械、tRNA测序不仅可能成为一种微生物反应获得洞察细微的环境变化,不能轻松地以其他手段来衡量,但也带来更多的生物学和RNA RNA表观遗传学到微生物的快速发展的领域。”
研究人员对他们的发现感到兴奋,但也认为这种新策略还有很大的改进空间,他们希望这种新策略能很快实现。
“有很多方法可以检测微生物组的活动,但没有什么比测序更快、数据量更大的了,”潘博士总结道。“在这里,我们开发了一种新的方法,可以通过tRNA报告微生物组的活动,并以高通量的方式进行。这才是真正的价值所在。
推荐内容
-
河马常常张大嘴巴打哈欠其实是?蚂蚁庄园9月19日今日课堂答案
2020年9月19日,今天是周六,蚂蚁庄园小课堂的问题更新了,今天的问题还是比较有意思的,就是河马常常张大嘴巴打哈欠其实是?人们从以下哪种动
-
6月17日上海疫情最新数据情况公布 上海新增境外输入确诊病例2例
【上海疫情】抗疫之战已持续一年之久,从当前的环境来看,疫情虽然已经得到了很好的控制,但也可能在局部地区进一步扩散。为此,希望大...
-
11月5日大连市疫情数据公布 昨日,大连新增8例本土确诊病例
温馨提示:尚未接种新冠病毒疫苗的群众(有接种禁忌症除外),请尽快持本人身份证到附近的接种点完成疫苗接种。继续加强个人防护意识,养...
-
索尔克的科学家们逆转了衰老的迹象
白发苍苍,鱼尾纹斑驳,与20岁时相比,如今的伤口需要更长的时间才能痊愈。面对衰老的明显迹象,我们大多数人至少有过一次让时光倒流的...
-
心理困扰是痴呆症的风险因素
一项新的研究表明,重要的疲惫 - 可以被视为心理困扰的指标 - 是未来痴呆风险的一个风险因素。哥本哈根大学公共卫生系的研究人员与国家
-
中国生物:牛痘已安全封存以备不时之需
中国生物:牛痘已安全封存以备不时之需 原标题:中国生物:牛痘已安全封存以备不时之需
-
糖尿病为什么要用动态血糖仪器监测血糖?
糖尿病为什么要用动态血糖仪器监测血糖? 原标题:糖尿病为什么要用动态血糖仪器监测血糖?
-
1月25日北京市大兴区疫情最新消息 北京一确诊病例曾三次检测呈阴性
【导读】在春运即将启动之际,希望广大民众积极配合防疫工作的展开,相信以全中国人民的力量,我们定能一同渡过这次难关。同时,目前,...
-
何猷君回应改认证 何猷君回应说了什么?
赌王何鸿燊去世之后,何猷君一直备受争议,先是发了一家四口的合照,后又更改了身份认证,一度引发了网友的广泛质疑,甚至在网上成为了热搜话
-
苏州市相城区关于4月25日在部分区域开展核酸检测的公告
苏州市相城区关于4月25日在部分区域开展核酸检测的公告 原标题:苏州市相城区关于4月25日在部分区域开展核