准确的DNA拼写校正方法
基础科学研究所(IBS)的研究人员证明了最近开发的基因编辑方法的准确性。这就像“DNA剪刀”一样,旨在识别和替代30亿中的一个核苷酸。“这是第一次在整个基因组水平上验证该基础编辑器的准确性,”该研究的主要作者KIM Jin-Soo解释说。该研究发表在Nature Biotechnology上,有助于扩大该方法在农业,畜牧业和基因治疗中的应用。
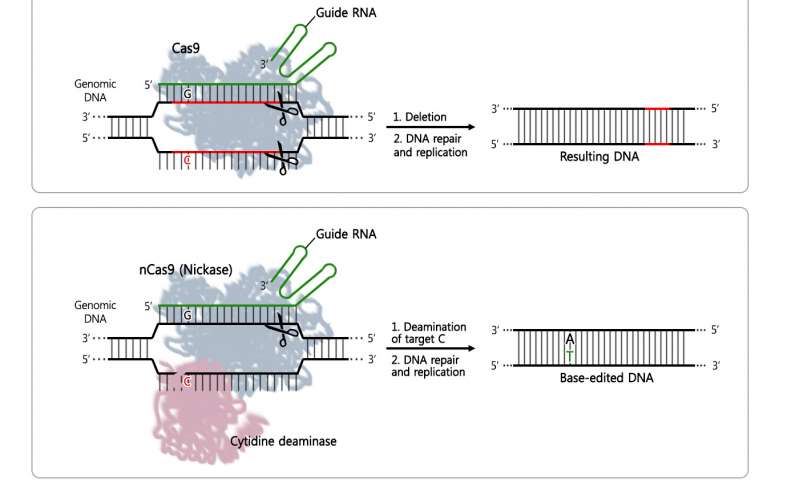
基因编辑工具的快速发展为生物界带来了兴奋。主要的第三代DNA编辑技术是CRISPR - 一种比其前辈更快,更便宜的工具。通过切除小DNA序列,CRISPR-Cas9和CRISPR-Cpf1用于沉默或减少缺陷基因的表达。然而,去年,生物学家发现了一种新的碱基编辑方法,它不会导致随机DNA缺失和插入,而是只替换一个DNA碱基。这些类型的基因校正是至关重要的,因为几种疾病是由DNA四种基本成分之一的拼写引起的;腺嘌呤(A),胞嘧啶(C),鸟嘌呤(G)和胸腺嘧啶(T)。DNA中的单核苷酸错误称为点突变。由点突变引起的病症的实例包括囊性纤维化,镰状细胞性贫血和色盲。
与现有技术不同,基础编辑器方法包括与另一种称为胞嘧啶脱氨酶的酶融合的CRISPR-Cas9(nCas9,切口酶)的变体,其用T代替DNA组分C.剪刀指向DNA上的正确位置通过指导RNA。然而,直到最近,还不知道基础编辑是否仅在有缺陷的基因区域中起作用,或者是否在不合适的区域中不必要地替换了Cs。
报告在Nature Biotechnology中首次成功进行基因编辑以修饰肌营养不良蛋白和酪氨酸酶基因中的单个核苷酸仅一个月后,该团队在基因组规模上证明了该方法的准确性。
为了确定方法的完整性,IBS研究人员修改了称为Digenome-seq的错误检查技术,以使其适应基本编辑器方法。Digenome-seq去年被用于验证,当时该团队分析了CRISPR-Cpf1和Cas9的准确性。IBS研究人员还改进了计算机程序Digenome 2.0,以更全面地识别脱靶,并比较不同的指导RNA,以找到减少故障和提高特异性的指南。
使用这种技术,该团队发现基础编辑技术比当前的第三代CRISPR-Cas9更精确。基础编辑技术在人类基因组的多个位点诱导C-to-T转换,而CRISPR-Cas9在多个位点引起切割,这意味着新的基础编辑器技术减少了脱靶变化。“因此,预计这些基础编辑器将像流行的CRISPR技术一样广泛使用,”KIM说。
推荐内容
-
多样性和移民提高了微生物群落的生产力
新的研究显示,自然选择很快将微生物的大熔炉变成了一个高效的社区。埃克塞特大学的科学家将十个不同的产甲烷群落(数百种微生物种群,主要
-
研究人员报告了护送一种关键酶的蛋白质用于细胞分化
成体生物的发育始于受精卵,其分化成数百种包含组织和器官的特化细胞类型。这些细胞命运如何发生变化是研究兴趣的主题。所有细胞都含有...
-
快速鉴定疟疾基因功能的新方法
来自沙特阿拉伯,日本和英国的研究团队之间的国际合作使得研究人员在开始实验后的14到18周内将基因与其在疟疾寄生虫中的功能联系起来。了解
-
钻石黑范氏贫血发病机理中的信号通路
Diamond Blackfan Anemia(DBA)是一种以骨髓无法产生红血球,先天性异常和易患癌症为特征的疾病。当前的治疗选择,包括类固醇治疗和慢性输
-
瘟疫细菌在变形虫中避难
鼠疫耶尔森氏菌(Yersinia pestis)是引起腺鼠疫的细菌,它可以在无处不在的土壤原生动物变形虫中生存,通过产生保护免受后者微生物消化的蛋
-
哥伦比亚大学的研究人员揭开了为什么有些胶质母细胞瘤对免疫疗法
哥伦比亚大学的研究人员已经了解了为什么一些胶质母细胞瘤 - 最常见的脑癌类型 - 对免疫治疗有反应。该研究结果有助于确定最有可能从免
-
认知的轻微下降预示着阿尔茨海默氏病的进展
阿尔茨海默氏病(AD)是进行性的,但发展缓慢-或至少无法自我显示。在一项于2020年2月14日在线发表在《生物精神病学》杂志上的新研究中,加利
-
科学家发现土壤细菌需要两层安全就像数字世界一样
谷歌的那些人认为他们太聪明了。苹果和微软也是如此。他们的软件(和许多其他人)在数字世界中使用双因素身份验证来验证身份,但他们有点...
-
益生菌还可以减少局部糖皮质激素的需求
JAMA Dermatology上发表的一项研究表明,每天服用一粒益生菌鸡尾酒胶囊可以减轻中度特应性皮炎的严重症状,也可以减少局部皮质类固醇激素
-
丹纳赫将以21.4亿美元的价格收购GE生命科学公司的生物制药业务
丹纳赫今天表示已同意以约214亿美元的现金收购GE生命科学的生物制药业务,该协议旨在增强买方的生物加工产品并偿还卖方的债务。丹纳赫表示