研究人员创造了细胞ORACL以帮助药物发现
加利福尼亚大学旧金山分校的一个研究小组设计了一种新的早期药物发现方法,该方法利用计算机视觉领域的技术与强大的新工具相结合:一种改变其外观的转基因癌细胞谱系用针对常见疾病途径的药物治疗。研究人员希望利用这些细胞 - 结合类似于人脸识别软件所使用的算法 - 根据其生物学功能快速组织机构化合物库中数十万个分子,极大地促进了对新药和更好药物的研究。用于治疗癌症和其他疾病。出于这个原因,他们将细胞称为ORACLs(Optimized Reporter细胞系,用于注释复合文库)。
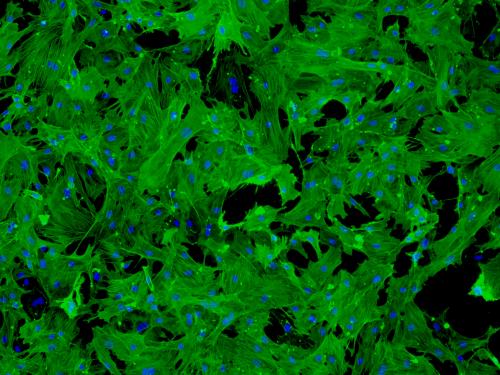
描述新方法的论文于2015年12月14日在线发表在Nature Biotechnology杂志上。
共同资深作者Steven Altschuler博士和Lani Wu博士是加州大学旧金山分校药学院的药物化学教授,并在德克萨斯大学西南医学中心进行了该领域的初步研究。他们认为药物发现更像是基因组学领域,研究人员花费数十年时间对大型基因组数据库进行注释,因为越来越多的功能被发现用于个体基因和调控元素。通过这种社区努力,研究人员现在可以快速识别感兴趣的特定基因,以进行更有针对性的调查。
使用ORACL对现有化合物库进行功能性注释可以获得类似的好处,使研究人员能够快速鉴定与特定疾病途径临床相关的药物,并显着简化科学家使用共享药物发现设施或核心的方式,这在学术研究中很常见。
“药物筛选核心非常繁忙 - 它们不断运行,”吴说。“问题在于筛查结果通常无法重复使用。当你有一个新的生物靶点,你想用药物打击时,你必须再次筛选整个化合物库。”
形状转换细胞揭示了药物机制
研究人员通常使用“报告细胞”筛选有效的药物,这些细胞经过精心设计,可以改变其行为或外观以应对成功治疗。但是,ORACL的创建 - 一种能够区分多种常见药物类别的单一报告细胞系 - 需要采用不同的方法。
“我们不知道如何根据我们现有的生物学知识设计这样一个多功能的记者细胞,”Chien-Hsiang“Charles”Hsu说,他是Altschuler和吴实验室的研究生,也是新研究的共同主要作者。“所以我们想,为什么不随机标记蛋白质并用药物筛选细胞,看看哪条生产线可以产生符合我们目标的反应?”
研究人员根据现有的肺癌细胞系,通过用荧光标记标记随机选择的基因,创建了93种细胞系。他们用一组属于六种常用癌症药物的30种化合物处理来自这些谱系的细胞,然后用自定义算法分析细胞图像。正如团队所希望的那样,不同的药物使细胞以不同的方式改变其形状和荧光模式,使软件能够推断出应用了哪种类型的药物。
正如预期的那样,一些细胞系比其他细胞系更具信息性:研究人员高兴的是,一种细胞系证明能够区分六种药物类别,准确率为94%。细胞对不同药物类型的特征反应的一些更微妙的方面需要计算机算法检测,但在显微镜下其他一些是显而易见的:DNA损伤药物导致细胞膨胀,而HDAC抑制剂产生尖锐的外观。MTOR抑制剂在整个细胞中产生暗淡的荧光,而用hsp90抑制剂处理导致细胞质内的荧光斑点。
研究人员发现了他们的ORACL。
Oracular细胞可以筛选化合物库以查找被忽视的药物
为了测试他们的ORACL细胞,该团队筛选了来自几个机构化合物库的10,000多个未知功能的小分子。他们的分析鉴定了106个分子,这些分子对细胞的影响与最初选择ORACL的“训练”药物相匹配,进一步的实验证实,这些匹配中至少有90个影响与训练药物相同的生物途径。
令研究人员惊讶的是,ORACL细胞也对一系列其他化合物产生了特征性反应,其中一些化合物属于原始训练集中未包括的药物类别,包括ER和Aurora激酶抑制剂,糖皮质激素类固醇,ATP酶抑制剂,和二氢叶酸还原酶抑制剂,以及其他十几种分子,其共同的生物学功能尚不清楚。“令人惊奇的是,”Altschuler说,“我们能够一次完成一个屏幕,同时捕捉到许多不同类别的分子。”
目前,研究人员正在扩大其筛选范围,以便快速注释含有数十万个分子的化合物库。他们希望采用ORACL来识别新化合物,这些化合物会影响生物学途径,目前的药物有太多的副作用,或者错过某些患者群体。他们可以找到影响由少数已知药物或甚至药物影响的途径的化合物,其效果似乎远离任何已知的生物途径。“这些都是药物发现的早期步骤,”Altschuler说。“我们希望找到更多高质量的化合物将使所有后续步骤更加高效。目前这个过程需要花费数十亿美元多年。这样做会不会更好?”
推荐内容
-
明确了!常态化核酸检测费用,各地政府承担
明确了!常态化核酸检测费用,各地政府承担 (来源:玉溪中院) 原标题:明确了!常
-
7月24日越南胡志明疫情最新数据公布 越南新增新冠确诊7307例
所谓后疫情时代,并不是我们原来想象的疫情完全消失,一切恢复如前的状况,而是疫情时起时伏,随时都可能小规模爆发。据消息显示,越南...
-
搜狐医药 | 上海:16个区都已全面实现社会面清零
搜狐医药 | 上海:16个区都已全面实现社会面清零 原标题:搜狐医药 | 上海:16个区都已全面实现社会面
-
为什么美元暴跌黄金跟着跌 黄金暴跌的深层原因
在很多人的想法中,美元下跌的情况下,黄金就一定会是上涨的态势。但是这一周发生的状况就让人蒙蔽了。美股史无前例的暴跌!一周连续两次向
-
中风的原因有哪些? 为什么会中风?
中风从它的病因来说,主要可以分为5大类:第一类就是动脉粥样硬化,我们知道有高脂血症等这样的情况下,在长期的过程中,血管壁可以发生动
-
美国医疗保健支出在发达国家中最高
按人均计算,美国在医疗保健方面的支出比其他发达国家要多得多; 根据约翰霍普金斯大学彭博公共卫生学院研究员领导的研究小组的研究,主要
-
人工智能可以大大减少处理异常胸部X射线所需的时间
新的研究发现,一种新颖的人工智能(AI)系统可以大大减少确保具有关键发现的异常胸部X射线将更快获得专家放射学家意见所需的时间,将平均延
-
疫情之下,住在养老院的老人自我保护要做好这两点
疫情之下,住在养老院的老人自我保护要做好这两点 原标题:疫情之下,住在养老院的老人自我保护要做好这两
-
犬特征的多样性归因于简单遗传结构
在任何一天都可以进入狗公园,你一定会看到狗在比赛中的大小,形状和颜色的显着差异。家养犬的体型大小与任何哺乳动物物种的差异最大。...
-
教育是场持久战 你坚持住了吗?
中国有句话大家都常挂在嘴上:活到老学到老。当前一场疫情下来,很多家长全然没有了居家度假的喜悦,个个被同样禁足在家里的孩子缠得头...






