研究人员揭示了由胞嘧啶碱基编辑引起的意外全基因组脱靶突变
中国科学家发现胞嘧啶碱基编辑器(BE3和HF1-BE3)诱导全基因组脱靶突变。由中国科学院遗传与发育生物学研究所高彩霞教授领导的研究小组利用水稻全基因组测序(WGS)对BE3,HF1-BE3和ABE 的脱靶突变进行了全面调查。,一种重要的作物品种。
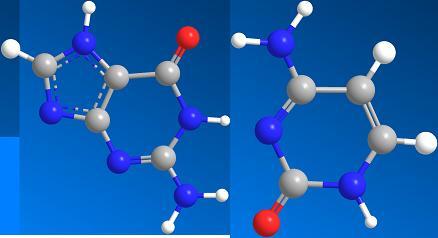
单核苷酸改变是人类疾病和经济生物中性状变异的重要原因。核碱基编码器对单核苷酸多态性的基因工程为基因治疗带来了巨大希望,它可以潜在地治愈人类疾病并改善作物植物的性状。
研究人员已经开发出胞嘧啶和腺嘌呤碱基编辑器(分别是CBE和ABE)。基础编辑器是切口酶型Cas9蛋白与胞嘧啶脱氨酶或腺嘌呤脱氨酶的融合,并且已经发现催化单个指导RNA(sgRNA)的靶位点中C> T或A> G的转化。 。
尽管CBE和ABE的靶向转化在许多生物体中发现,但它们的脱靶效应尚未在全基因组水平上进行系统评估。
基础编辑特异性的过去分析主要局限于使用计算机软件预测的类似靶标的位点。这些类似靶标的位点通常数量少并且基因组分布受限。
考虑到大肠杆菌,酵母细胞和人类细胞中胞嘧啶脱氨酶的异位表达已经被发现引发全基因组脱氨基因事件,在全基因组水平和检测基因编辑器的特异性已经变得必要和迫切。无偏见的方式。
研究人员选择了三种广泛使用的基础编辑器:BE3,高保真BE3(HF1-BE3)和ABE。将靶向11个基因组位点的总共14个碱基编辑构建体通过农杆菌转化转化到水稻中。
由BE3,HF1-BE3或ABE编辑的再生T0植物和用基础编辑但没有sgRNA转化的植物,以及两个对照组的植物(即野生型水稻和转基因水稻的无效分离株),由WGS分析。
基础编辑器组和控制组在找到的插入数量上没有显着差异。相反,BE3和HF1-BE3组具有比ABE和对照组显着更多的单核苷酸变体(SNV)。
每株植物的C> T单核苷酸变体(SNV)的平均数为:203(BE3); 347(HF1-BE3); 88(ABE); 和105(对照组)。因此,BE3和HF1-BE3植物中的C> T SNV分别比对照植物高94.5%和231.9%。
值得注意的是,在没有sgRNA的情况下用BE3和HF1-BE3处理水稻植物也导致大量的C> T SNV。此外,由BE3和HF1-BE3赋予的绝大多数额外C> T突变与使用计算机软件(Cas-OFFinder)预测的脱靶位点不匹配。
发现所有SNV以及C> T SNV分布在整个水稻基因组中,表明全基因组发生。用转录组数据作图显示,与转录的基因区域相比,与BE3和HF1-BE3相关的大量C> T SNV更频繁地发生,其中由于活性转录而产生单链DNA。
总而言之,高博士团队生成的数据表明,BE3和HF1-BE3(而非ABE)在水稻中诱导全基因组脱靶突变。这些脱靶突变,主要是C> T SNV并且在转录的基因区域中富集,不是通过当前的计算机方法预测的。含有胞嘧啶脱氨酶的碱基转化单元可能是BE3和HF1-BE3引发的大量脱靶SNV的原因,需要进行优化以提高胞嘧啶碱基编辑的特异性。
“基因编辑器代表了一种吸引人的工具,用于产生植物育种所需的精确遗传变异。这些编辑的特异性至关重要,因为脱靶突变可能是有害的。我们发现当前的BE3或HF1-BE3导致意外和不可预测的基因组植物中广泛的脱靶突变,凸显了优化这类编辑特异性的紧迫性,“高博士说。
推荐内容
-
用铁锅炒菜就能补上身体所缺的铁吗?听听专家怎么说
互联网时代,每个人都是信息传播者,但是我们必须清楚一点:在没有获得真正证实的消息之前,千万不能随意散播谣言,很容易造成以讹传讹...
-
NHGRI研究人员和合作者在不同的人群中识别出Noonan综合症
美国国立卫生研究院的国家人类基因组研究所(NHGRI)及其合作者的研究人员已经成功地使用面部分析软件来诊断非洲人,亚洲人和拉丁美洲人的Noo
-
转基因甜玉米不再对戈斯的枯萎病敏感
表达对除草剂草甘膦(GR)的抗性的转基因作物已经商业化并在美国广泛种植了二十年。大多数转基因玉米(Bt)也被设计用于产生对某些玉米害虫有效
-
引起疾病的胃虫攻击宿主细胞中的能量产生
研究人员在一项新的研究报告中指出,幽门螺杆菌- 胃炎,溃疡和胃癌的主要原因 - 通过关闭胃壁细胞内的能量产生来抵抗感染的屏障,从而抵
-
怀孕到底能不能养猫狗?其实真相是...
生活中我们经常会听见各种各样的流言,但是只要我们用心去思考和辨认其实 不难发现这些谣言有很多破绽。所以今天,小编照常给大家辟谣一则
-
男人吃西瓜等于
-2022年4月16日发(作者:海狗鞭壮阳吗)男人吃西瓜等于?食疗不等于药补中国人大多坚信“药补不如食补”,因为他们觉得,食物不会像化学...
-
利用“过午不食”法养生?来看学者的建议
互联网时代,每个人都是信息传播者,但是我们必须清楚一点:在没有获得真正证实的消息之前,千万不能随意散播谣言,很容易造成以讹传讹...
-
吃壮阳药对身体的危害是什么?
-2022年4月16日发(作者:石花菜)如对您有帮助,可购买打赏,谢谢吃壮阳药对身体的危害是什么?导语:现在很多人的夫妻生活都不是特别的圆满
-
科普孩子营养不良的5个表现及胡萝卜的营养功效
有些孩子会挑食、厌食,家长会以为是孩子没有养成良好的吃饭习惯,其实还有可能是缺锌。缺锌会让孩子基本上对所有食物都不感兴趣,严重...
-
每天喝4升水可能会中毒?你了解多少
当我们不知道一件事真假的时候,不防多等等官方的回应。因为信息碎片化,很多小伙伴现在上网很容易只看到事情的一面就下定论,其实很多...