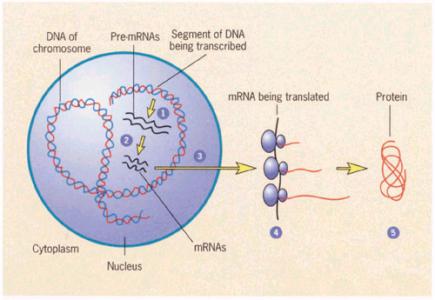
RNA序列排列显示精子微生物群
一项新的研究报告了人类精子微生物群的首次详细描述,使用了一种新的RNA,该RNA排列了能够区分精子RNA和细菌污染或定植的技术。

这项研究发表在2020年1月的《生殖与遗传学》杂志上。研究表明,安排非目标RNA序列有可能识别感染的存在,以及检测精子的存在。因此,在对不孕不育人群进行常规评估期间,这可能为更好和更个性化的护理提供机会。
RNA排序是一种遗传技术,在这种技术中,RNA的数量和序列被发现使用下一代排序(NGS)技术。它基本上是对转录组的探索,是所有转录的dna RNA的总和,包括mRNA、tRNA和rRNA。这有助于将dna信息,或基因表达,与真正合成的蛋白质联系起来,这些蛋白质决定了细胞的真正功能。它显示了正在分析的样本中的基因活动水平——哪些基因实际上正在被转录,在什么水平上,在什么时期的堆栈活动。这有助于发现细胞是如何工作的,哪些蛋白质在不同的阶段和不同的目的变得活跃,以及伴随或先于疾病条件的一些变化。有许多基于RNA序列排列的技术,包括转录谱、SNP鉴定、RNA编辑和遗传表达的差异分析。
排列RNA序列比排列dna序列更能捕捉基因数据的真实表达方式。例如,它可以通过显示一个未知的基因在哪些组织中是活跃的来帮助理解它的作用。它展示了一个基因如何通过替代修饰等机制导致两个或两个以上不同的RNA序列,这是一个不可能安排dna序列的发现。它还可以展示RNA是如何被加工成具有不同功能的分子的,比如添加聚腺苷链或5"缓冲液。
最初基于桑格排序,RNA排序缓慢、昂贵且不确定——尽管桑格的方法在当时是一项不可思议的创新。只有随着NGS等新技术的出现,RNA序列的排列才变得切实可行,而且非常有用。
NGS是多生产高产的保护伞术语,它排列序列技术可以很容易、准确和廉价地排列dna和RNA序列。
这项研究是在韦恩州立大学医学院、生育中心和马萨诸塞大学阿姆赫斯特分校的合作下进行的。
这项研究的目的是发现,将非目标RNA序列排列成足够敏感和特异性的序列,以检测微生物的存在,而目前使用目标培养的技术无法做到这一点。研究人员采集了85个精液样本,并从精子中提取了RNA。这是按顺序排列的,然后使用它的增强技术。不属于人类基因组的RNA序列与微生物基因结合。这些读数被用来创建在每个精液样本中发现的微生物的图像。
他们发现,所有的样本都显示出正常情况下在男性生殖范围内发现的细菌水平相似。这些来自11属,4行。只有一个例外,它与所有剩余的微生物读数有很大的不同。
具体地说,这个标本中有大量的乳酸链球菌无乳杆菌和该物种的链球菌。S.无乳杆菌是一种细菌,与许多新生儿感染以及妊娠和分娩后发生的感染有关。它也是许多早产婴儿死亡的原因。此外,它还会导致老年成人患者的严重感染。因此,精子中这种细菌的存在引起了相当大的兴趣。
目前,男性生殖器官中微生物的存在是通过培养技术来诊断的。然而,大多数通常被发现污染这一范围的微生物在培养要求上都很挑剔,这意味着它们不能在通常的实验室条件下培养。
另一方面,安排RNA序列是一种更安全的技术,现在已经得到了更广泛的应用,而且每天都在变得越来越普遍。这项技术的使用可能会更好地了解居住在人体中的细菌和其他微生物。研究人员说:“我们已经证明,将人类精子RNA的非目标序列排列起来,有可能提供微生物(细菌、病毒、古细菌)的特征。”这些信息通常是从侧面成型的数据中恢复的,作为常规核酸排列顺序的一部分。
临床图像显示,S.无乳症感染呈上升趋势,比以往任何时候都更加严重。其他微生物也在新生儿和成人中引起更频繁的感染。在这种情况下,研究人员斯蒂芬·克拉韦茨(Stephen Krawetz)说,“人类非目标精子RNA排列数据序列,除了提供生育状态外,还可以作为微生物状态的诊断工具。”
推荐内容
-
人工甜味剂阿斯巴甜会致癌吗?具体什么情况呢
生活中我们经常会听见各种各样的流言,但是只要我们用心去思考和辨认其实 不难发现这些谣言有很多破绽。所以今天,小编照常给大家辟谣一则
-
T细胞转录因子可为疫苗研究提供新途径
成功疫苗的黄金标准是产生长效抗体,多年来可以保护身体免受疾病侵害。现在,由国家人类基因组研究所(NHGRI)的科学家领导的研究小组已经为
-
人类基因组计划启动后的四分之一世纪超越基础对的经验教训
2015年10月1日,标志着25 日推出的人类基因组计划的周年纪念日。为了纪念这一周年,展示人类基因组计划的丰富的历史和基因组学在过去的四
-
罕见的致死性疾病可追溯到X染色体上PIGA基因的变异
包括约翰斯·霍普金斯大学和儿童国家医学中心同事在内的临床和实验室研究人员团队使用下一代基因组测序,除了经典生物学,还可以找到导...
-
鲸眼的基因突变可能会增加死亡风险
科学家们发现,露脊鲸眼中的基因突变妨碍了他们在明亮的光线下观察的能力,这可能使他们更容易受到渔具致命缠绕的影响,渔具是这种极度...
-
隔着玻璃晒太阳:?来听听专业人士怎么说 只要时间够长,就能有
随着互联网的快速发展,我们在获取信息的时候很容易被带跑方向。有时候真相也许并不是网上说的那样,所以我们必须要有自己的判断和独立...
-
研究人员发现在蛋白质合成过程中形成机械力的特征
像任何装配线一样,身体的蛋白质构建过程产生机械力,因为它产生这些重要的细胞构建块。现在,一组研究人员表示他们距离理解这种力量还...
-
怎么吃西红柿?真实情况是怎样 才能发挥其预防前列腺癌的作用?
随着互联网的快速发展,我们在获取信息的时候很容易被带跑方向。有时候真相也许并不是网上说的那样,所以我们必须要有自己的判断和独立...
-
国家卫健委:我国全程接种人数占全国总人口的88.11% 鼓励老年朋友
今日,国务院联防联控机制召开新闻发布会,介绍从严从实抓好疫情防控工作有关情况,并答记者问。 接种新冠疫苗获益要大大高于风险 中国
-
你需要用营养素补充剂替代食物吗?来看学者的建议
生活中我们经常会听见各种各样的流言,但是只要我们用心去思考和辨认其实 不难发现这些谣言有很多破绽。所以今天,小编照常给大家辟谣一则