游戏玩家和AI的结合帮助改善了细胞和蛋白质的定位
在一张显示数十万人体细胞微观图像的地图上,一个国际研究团队正在与游戏社区和人工智能合作,以更细致地了解细胞内排列的蛋白质模式。
KTH皇家理工学院,CCP Games和Massively Multiplayer Online Science合作报道了这些进展。
该研究发表在9月出版的Nature Biotechnology上。研究人员报告称,游戏玩家或“公民科学家”推动了用于在亚细胞水平上预测蛋白质定位的AI系统。众包和AI的结合改善了亚细胞蛋白质模式的分类,并首次鉴定了被称为“棒和环”的细胞结构家族的10个新成员,来自KTH的研究员Emma Lundberg说道。 Atlas,人类蛋白质图谱的一部分,在Science for Life联合研究中心。
伦德伯格表示,该数据正在积极整合到公众可用的人类蛋白质图谱数据库中,并将成为全球研究人员的资源,致力于更好地了解人类细胞,蛋白质和疾病的发展。
研究人员与Massively Multiplayer Online Science和CCP Games合作,将人类蛋白质图谱中的蛋白质定位分析直接整合到EVE Online中,这是一款流行的大型多人在线游戏。由此产生的迷你游戏,名为“项目发现”,以伦德伯格的化身为特色,使她成为第一个在网络游戏中出现的活科学家之一。它由EVE Online的30多万公民科学家共同发挥,他们共同创造了超过3300万个蛋白质亚细胞定位的图像分类,这一成就被誉为公民科学的里程碑。
将公民科学家的能力与用于从图像中预测蛋白质亚细胞定位的AI系统进行比较,称为定位细胞注释工具(Loc-CAT)。Loc-CAT是第一个通过图像多重定位来注释蛋白质并在大量细胞类型中进行推广的通用工具,为研究细胞及其未来行为提供了有用的工具。
尽管Loc-CAT在许多常见类别的蛋白质中都优于Project Discovery的玩家,但来自EVE Online的聚合玩家数据更好地识别了稀有类别,并且能够注释训练数据不可用的新模式。通过将玩家的注释与机器学习方法相结合,转移学习被用来显着提高Loc-CAT的性能。
“我相信将科学任务整合到已有的计算机游戏中将成为未来利用人类大脑处理能力的常用方法,并且直接投入机器学习模型的公民科学游戏的复杂设计具有快速的能力利用大规模科学努力的成果,“伦德伯格说。“我们感谢参与这个项目的所有公民科学家,以及他们所做的发现。”
尽管这项工作取得了成功,但仍有很大的改进空间。研究人员从9月17日开始宣布人类蛋白质Atlas 2018挑战Kaggle(www.kaggle.com/competitions)。挑战将涉及图像分析,以分类人类细胞中的亚细胞蛋白质模式。数以千计的奖金将用于争夺,参与者的贡献将有助于推动蛋白质生物学领域的发展。
推荐内容
-
新郎带队到新娘家接亲 到门口一看暴怒扔掉捧花转身就走引热议
12月1日,贵州一位新郎大婚当天带队去新娘家接亲,到对方家门口一看却突然暴怒,当场爆粗口扔掉捧花转身就走,原因曝光引发热议,具体为什
-
辍学为父追凶17年男子发声 向明钱目睹父亲被杀是谁酿成的悲剧?
今日辍学为父追凶17年男子发声登录了热搜,也是在网上引起了网友们的关注,9岁男孩为父寻仇追凶17年,牺牲一生中最宝贵的时光去寻找凶手,
-
今日广东疫情最新数据公布 广州本轮疫情重症患者清零
【导读】抗疫之战已持续一年之久,从当前的环境来看,疫情虽然已经得到了很好的控制,但也可能在局部地区进一步扩散。为此,希望大家不...
-
文山王华聪最新消息 云南亿万富翁被杀案一审宣判结果如何?
近来关于文山王华聪最新消息传来了,今日云南亿万富翁被杀案一审宣判,那么案件的结果如何呢?此事已经过去两年了那么对于王华聪马寿兴被杀
-
12月3日内蒙古满洲里隔离区疫情消息公布 满洲里市已启用15个隔离
满洲里,由内蒙古自治区呼伦贝尔市代管,是内蒙古自治区计划单列市,是中国最大的陆运口岸城市,是国务院确定的国家重点开发开放试验区...
-
8月9日荆门高新区·掇刀区疫情最新数据公布 荆门掇刀区昨日新
温馨提示:一旦出现发热、干咳、乏力、鼻塞、流涕、咽痛、嗅觉味觉减退、结膜炎、肌痛和腹泻等症状,应及时按规范程序就诊,并主动告知1...
-
上海电气总裁黄瓯去世的原因是什么?上海电气黄瓯家庭背景如何资
今天发生一个令人悲痛的消息,上海电气黄瓯突然离世,关于黄瓯简历也备受关注。而对于上海电气总裁黄瓯离世的原因,想必大家也是非常疑...
-
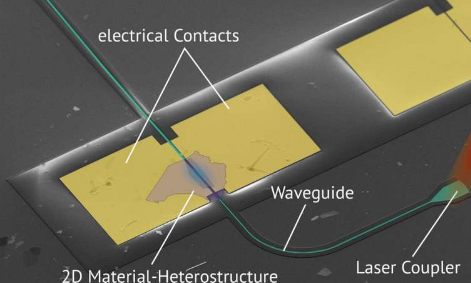
由二维材料制成的快速光检测器
苏黎世联邦理工学院的两个研究小组已经联合开发了一种新型的光探测器。它由耦合到硅光波导的不同材料的二维层组成。将来,这种方法也可...
-
12月2日云南德宏州疫情最新数据公布 云南昨日新增境外输入确诊3例
云南12月1日新增境外输入确诊3例、无症状感染者1例。据消息显示,云南卫健委12月2日通报,12月1日0-24时,全省新增确诊病例3例,均为境外输
-
11月16日成都理工大学疫情最新消息公布 从即日起,校园封闭
成都理工大学学生宿舍今天9时起解除封闭管理。那么,对于成都理工大学疫情的这个话题,今天的你是否也在关注着?具体什么情况?下面跟小编一