研究人员确定了转化染色体的分子运动
麻省理工学院和法国巴斯德研究所的研究人员已经通过形成DNA环来将基因组组织成不同邻域的分子“运动”。在2016年发表的一项研究中,由麻省理工学院医学工程与科学研究所物理学教授Leonid Mirny领导的研究小组提出,分子马达将染色体从松散缠结的状态转变为动态的一系列扩张环。
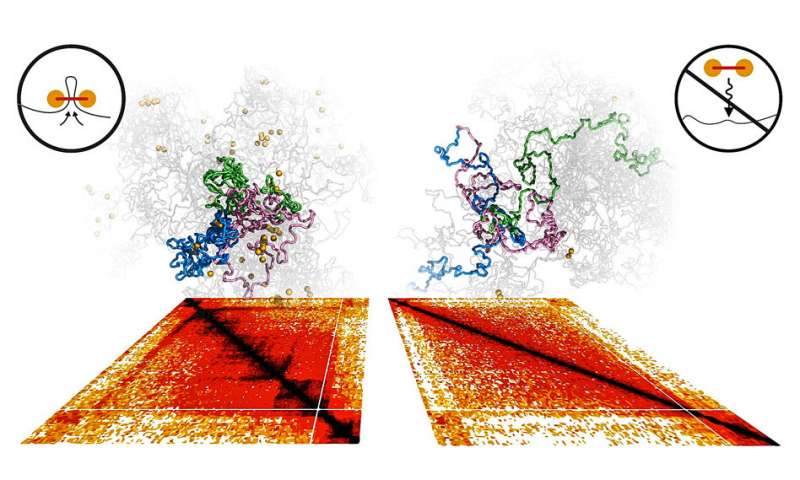
这种被称为环状挤出的过程被认为将调节元素与它们控制的基因结合在一起。该团队还建议DNA装饰有障碍物 - 类似于停止标志 - 限制挤压过程。通过这种方式,循环挤出将染色体划分为单独的调节邻域,称为拓扑关联域(TAD)。
然而,虽然研究人员提出一种叫做cohesin的环状蛋白质复合物可能是这些分子马达的候选者,但这还未得到证实。现在,在自然杂志上发表的一篇论文中,由巴斯德研究所的Mirny和Francois Spitz领导的研究小组已经证明,cohesin确实在循环挤出过程中起到了马达的作用。
“这些机器中的每一台都落在DNA上并开始挤出环路,但这些电机无法通过DNA界限,”Mirny说。“因此,由于这种运动活动,基因组被组织成许多不跨越边界的动态环,因此基因组被分成一系列邻域。”
研究人员还发现,一种不使用cohesin的不同机制正在将DNA的活性和非活性区域组织到细胞核中的独立区室中。
为了确定cohesin在基因组形成中所起的作用,研究小组首先删除了一种称为Nipbl的分子,该分子负责将cohesin加载到DNA上。
然后他们使用称为Hi-C的实验技术,其中捕获并测序在3-D空间中彼此接近的DNA部分,以测量染色体上不同斑点之间的物理相互作用的频率。
这项技术由伍斯特马萨诸塞大学医学中心生物化学和分子药理学教授Job Dekker开创,此前曾用于证明TAD的存在。
该团队首先使用Hi-C技术评估染色体的组织,然后从小鼠中取出Nipbl分子。然后他们取出分子并再次进行相同的测量。
他们发现邻里几乎消失了。
然而,基因组的活性区域和非活性区域之间的区室化变得更加显着。
该团队认为,cohesin马达允许每个基因接触其调节元件,这些元件控制是否应该打开或关闭基因。
更重要的是,似乎cohesin马达被另一种蛋白质CTCF阻止,CTCF定义了每个邻域的边界。在Cell杂志最近的一项研究中,Mirny实验室与旧金山加利福尼亚大学和马萨诸塞大学医学院的研究人员合作证明,如果去除这种分界蛋白,邻域之间的边界就会消失,从而允许基因在一个社区与监管要素交谈,他们不应该在另一个社区谈话,并导致细胞中基因的错误调控。
“Cohesin是基因调控的核心,我们强调这是一种运动功能,所以它们(基因及其调控元素)不仅在空间中随机发现,而且它们通过这种运动活动聚集在一起, “米尔尼说。
Dekker表示,本文为细胞折叠染色体的机制提供了重要的新分子见解,他没有参与当前的研究。
“在这项工作中,Mirny和Spitz实验室将小鼠模型与基因组方法结合起来研究染色体折叠,以揭示加载cohesin复合物的机器对于TAD形成至关重要,”Dekker说。“从这个和另一个先前的研究中,一种分子机制正在形成,其中TAD通过cohesin和Nipbl依赖的染色质环挤出形成,其被CTCF结合的位点阻断。”
研究人员现在试图描述分子运动的缺失将如何影响基因调控。他们还进行计算机模拟,以确定在基因组经历分离成活性和非活性区室的独立过程的同时基于粘连蛋白的环状挤出是如何发生的。
“就像两位钢琴演奏者在同一架钢琴上演奏一样,”米尔尼实验室的博士生Nezar Abdennur说道,他与博士生Anton Goloborodko一起参加了这项研究。“他们互相干扰并施加约束,但他们在一起可以产生一种美妙的音乐。”
推荐内容
-
研究表明 直到现在被认为是非编码的环状RNA可以编码蛋白质
以色列和德国的一组科学家,由耶路撒冷希伯来大学的Sebastian Kadener教授领导,发现了一种用于环状RNA的蛋白质编码功能。这种RNA分子在脑
-
济南市政府将搬迁至先行区? 具体情况是什么?
济南市政府将搬迁至先行区? 具体情况是什么?全民自媒体时代,信息被大众获取的途径多种多样,但其中很多消息还没被证实真实性就大范围传
-
DNA计算机使智能药物更近一步
埃因霍温科技大学(TU e)的研究人员提出了一种使用DNA计算机控制药物输送到血液中的新方法。在Nature Communications杂志上,由生物医
-
依赖与安全感
现在的人们越来越物质化了,什么都要攀比,其实生活是自己的,过得开不开心只有自己知道。有时候我们真的不需要在意别人的眼光,做自己...
-
吃姜可以缓解痛经?真是如此?
面对流言,希望我们第一时间想到的是寻找官方回应。而不是着急着把流言传播出去,毕竟现在网络相关的法律法规都出台了,网络上传播谣言...
-
炒蔬菜都应该多加盐?来听听专业人士怎么说
当我们不知道一件事真假的时候,不防多等等官方的回应。因为信息碎片化,很多小伙伴现在上网很容易只看到事情的一面就下定论,其实很多...




