了解DNA是如何被选择性地标记为禁止使用标记的
并不是所有的基因组在任何时候都需要激活。如果不加控制,有些区域很容易以有问题的方式在基因组周围跳跃;另一些则是为特定细胞或特定时间需要关闭的基因编码。细胞控制这些遗传元素的一种方法是使用化学上的“禁止使用”标志。这种被称为DNA甲基化的化学信号,已知在不同的细胞类型或细胞发育的不同阶段存在差异,但细胞如何调节DNA甲基化标记的确切位置的细节仍不清楚。

索尔克大学研究植物的科学家们发现了一小类蛋白质,它们控制着基因组中DNA甲基化标记的添加位置。他们在基因调控这方面的工作与从正常发育到细胞缺陷和疾病的过程高度相关,这些过程可能分别由植物和/或人类中错误的DNA甲基化模式引起。他们的论文发表在2018年5月7日的《自然遗传学》杂志上。
“如果我们想了解DNA甲基化模式的差异会导致植物的发育缺陷,或者在人类疾病如癌症,我们需要了解基因组DNA甲基化是针对特定区域的在正常情况下,“沙克说朱莉法律助理教授,论文的资深作者。“到目前为止,能够以如此精确的方式控制甲基化的因素一直难以捉摸。”
Law研究的是一种容易生长的杂草,拟南芥,它是第一种完成基因组测序的植物。在接下来的几年里,包括劳在内的科学家们一直在努力描述和理解这种植物的DNA甲基化模式,这种模式会在不改变DNA编码本身的情况下影响基因活性。这个过程在植物和动物中是相似的,但是在拟南芥中研究DNA甲基化要容易得多,因为植物比动物更能忍受甲基化缺陷,在动物中,甲基化的全局变化往往是致命的。
Law感兴趣的是了解控制DNA甲基化的途径是如何被调控的,不仅是为了控制整体甲基化模式,而且是为了使个体区域的调控成为可能——这是在给定的生物体中产生不同的DNA甲基化模式的关键一步。
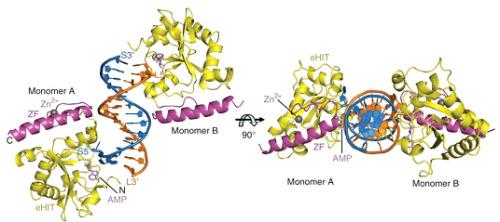
在此之前,已知一种名为RNA聚合酶IV (Pol-IV)的蛋白质复合物在建立DNA甲基化模式方面发挥着全球性的作用。这种聚合酶产生一种叫做siRNAs的小分子信息,其作用类似于分子GPS系统,指示基因组中甲基化应该靶向的所有位置。然而,这种聚合酶是如何被调节来控制DNA甲基化在个体基因组的位置尚不清楚。
为了解决这个问题,劳的实验室采用了一种基因-基因组结合的方法,研究了四种相关蛋白的功能,这四种蛋白被认为可能调节Pol-IV。结果发现,每一个上等基因的破坏都会导致不同的基因组区域——在不同的位置——失去它们的siRNA信号,导致DNA甲基化水平降低。更引人注目的是,当所有四种优质基因都被破坏时,siRNA信号和DNA甲基化在整个基因组中丢失。
索尔克研究所的研究员、论文的第一作者周明(音译)说:“在一流的四倍突变体中,Pol-IV信号完全消失了——基本上没有产生siRNAs。”“这是非常有力的证据,证明CLASSYs是Pol-IV函数所必需的。”
当Law的研究小组进一步探究时,他们发现,一流突变体中的DNA甲基化缺陷导致一些基因被错误地打开,导致移动DNA元素的甲基化在全球范围内下降,增加了它们四处移动的潜力,扰乱了基本的基因活动。
“CLASSYs是一个大型超家族的一部分,对于植物和动物来说都很常见,”Law补充说,他是赫斯特基金会发展主席。“我们希望通过了解特定的甲基化模式是如何在植物中产生的,我们可以对DNA甲基化在其他生物中是如何调控的提供见解。”
了解这种调节DNA甲基化的机制,可以帮助科学家开发出纠正表观遗传缺陷的策略。表观遗传缺陷与作物减产或疾病(如癌症)有关。在未来,该实验室的兴趣是探索DNA甲基化模式是如何在发展过程中控制和对环境的反应。
推荐内容
-
素媛案凶手居住地加装上百摄像头 监督人员增至4人
【素媛案凶手居住地加装上百摄像头 监督人员增至4人】想必大家在近期都有看到素媛案凶手将在今年年末出狱的消息吧!而对于他要出狱的情况,
-
海南将增加第四张离岛免税牌照 什么是离岛免税
【海南将增加第四张离岛免税牌照】关于海南将增加第四张离岛免税牌照这个网络上引起了网友的关注和热议,那么你知道海南离岛免税牌照是...
-
鲜奶比常温奶营养价值高?你知道吗
面对流言,希望我们第一时间想到的是寻找官方回应。而不是着急着把流言传播出去,毕竟现在网络相关的法律法规都出台了,网络上传播谣言...
-
研究中细胞的性别是否重要
在过去十年中,许多因毒性而从市场上撤出的药物被撤销,因为它们对女性的影响大于男性。事实证明,将药物推向市场的研究仅使用雄性细胞...
-
河南夏邑、虞城两县或合并,商丘有望晋升二线城市? 来看专业人
河南夏邑、虞城两县或合并,商丘有望晋升二线城市? 来看专业人士解答随着互联网时代的到来,信息传播速度越来越快,但其中很多消息还没被
-
服用维生素C补充剂能预防流感?其实是这样...
互联网时代,每个人都是信息传播者,但是我们必须清楚一点:在没有获得真正证实的消息之前,千万不能随意散播谣言,很容易造成以讹传讹...
-
研究人员发现COMMD3/8蛋白复合物是参与免疫细胞迁移的分子
由大阪大学免疫学前沿研究中心的Kazuhiro Suzuki领导的研究小组发现COMMD3 8复合物是一种参与免疫细胞迁移的分子,澄清该复合物在免疫
-
免疫规划成就宣传|设立专家咨询门诊助推预防接种工作高质量发展
免疫规划成就宣传|设立专家咨询门诊助推预防接种工作高质量发展 免疫规划成就宣传随着预防接种工作的深入开展和宣传工作的普及,广大