计算方法的速度可以帮助寻找新的抗生素
一个由美国和俄罗斯计算机科学家组成的团队已经开发出一种算法,可以快速搜索大量数据库,以发现已知抗生素的新变种 - 这是对抗抗生素抗性的潜在福音。
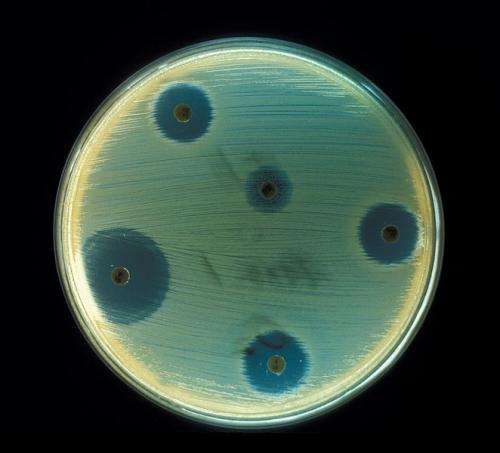
研究人员在最新一期的“自然微生物学”杂志上报告说,在短短几个小时内,该算法被称为VarQuest,其鉴定的肽天然产物(PNP)变体的数量是以前所有PNP发现工作的10倍。卡内基梅隆大学计算生物学系助理教授Hosein Mohimani表示,此前的搜索可能需要数百年的计算时间。
“我们的研究结果表明,微生物产生的抗生素比假设的多得多,”Mohimani说。他指出,VarQuest发现了超过一千种已知抗生素的变体,提供了微观生物学家在一次研究一种抗生素时无法获得的全局视角。
加州大学圣地亚哥分校计算机科学教授Mohimani和Pavel A. Pevzner设计并指导了这项工作,其中包括俄罗斯圣彼得堡国立大学的同事。
PNP在药理学方面具有无与伦比的记录。许多抗微生物和抗癌剂是PNP,包括所谓的“万不得已的抗生素”,万古霉素和达托霉素。随着对抗生素药物抗性的关注越来越多,找到更有效的已知抗生素变体是一种保持抗生素药物临床疗效的方法。
近年来,随着高通量方法的出现,对这些新型变体的研究得到了推动,这些方法可以批量处理环境样品,而不是一次处理一个。研究人员最近还推出了全球天然产物社会(GNPS)分子网络,这是一个由全球研究人员收集的天然产物质谱数据库。位于加州大学圣地亚哥分校的GNPS已经包含了超过10亿个质谱。
Mohimani说,GNPS代表了药物发现的金矿。他补充说,VarQuest算法采用更智能的方式对数据库进行索引以增强搜索,应该有助于GNPS实现其承诺。
Mohimani说:“自然产品发现正在变成大数据领域,该领域必须为收集,存储和理解大数据做好准备。”“VarQuest是消化社区已收集的大数据的第一步。”
推荐内容
-
心理知识:小孩子喜欢看鬼片是什么心理
日常生活中,我们会发现一个高情商的人是很受欢迎的。那么如何做一个情商高的人呢?其实只要懂得一点心理观察,善于捕捉一些细节,我们...
-
辟谣:隔夜茶真的致癌吗?真是如此?
当我们不知道一件事真假的时候,不防多等等官方的回应。因为信息碎片化,很多小伙伴现在上网很容易只看到事情的一面就下定论,其实很多...
-
心理知识:如何保持心理平衡_保持心理平衡妙招
心理这个词最近被提及的频率是越来越高了。其实在国外有一个职业特别吃香,那就是心理医生。为什么呢?因为国外特别是西方有几个国家已...
-
心理知识:什么人需要爱情障碍心理咨询
生活中,不管我们在跟自己独处还是跟别人相处的过程中其实我们会发现心理对一个人的影响是很大的。因此当我们发现自己有心理疾病的倾向...
-
哪些心理会影响受孕
心理这个词最近被提及的频率是越来越高了。其实在国外有一个职业特别吃香,那就是心理医生。为什么呢?因为国外特别是西方有几个国家已...
-
水中的DNA可以帮助揭示濒临灭绝的海牛生活的地方
尽管它们很大,但佛罗里达海牛并不总是那么容易找到。国家用于进行年度计数的航空调查因有时对笨重的海牛估计不足或过高而受到批评,并...
-
整体与部分的关系
心理这个词最近被提及的频率是越来越高了。其实在国外有一个职业特别吃香,那就是心理医生。为什么呢?因为国外特别是西方有几个国家已...
-
五谷杂粮打粉喝更好?其实是这样...
互联网时代,每个人都是信息传播者,但是我们必须清楚一点:在没有获得真正证实的消息之前,千万不能随意散播谣言,很容易造成以讹传讹...
-
小苏打被赶下神坛?大家都来看看 那些谣言你还信吗
随着互联网的快速发展,我们在获取信息的时候很容易被带跑方向。有时候真相也许并不是网上说的那样,所以我们必须要有自己的判断和独立...
-
每日形象 单细胞惊喜
英格尔细胞真核生物目前分成了门Hemimastigophora不愧是自己的超王国,代表只有10一个或更少的生命的真核生物树的最不同的分支,解释雅娜Eg