合成生物学家开发蛋白质语言的耳朵
就像字符串可以赋予含义一样,氨基酸序列可以赋予确定的三维结构以及所需的化学和生物学特性。这里的关键词是“可能”。在合成蛋白质中,氨基酸序列可能最终变得有意义或产生乱码。如何事先知道序列的“含义”?这个问题长期困扰着蛋白质工程师,他们寻求雄辩的,错误的,优雅的解决方案来解决生物制造问题。幸运的是,可能会有一个答案。这称为统一表示或UniRep(一种机器学习方法)。

UniRep来自哈佛大学的Wyss生物启发工程研究所,由乔治·丘奇(George Church)博士领导的研究小组利用一种人工智能的深度学习技术,直接从蛋白质的氨基酸序列中提取蛋白质的基本特征。根据丘奇和他的同事所说,这种方法不需要额外的信息,并且可以将许多费力的实验室实验转移到计算机上。
研究人员的深度学习方法于10月21日在《自然方法》上发表,文章标题为“ 基于序列的深度表示学习的统一理性蛋白质工程 ”。该文章指出,UniRep允许构建广泛适用和概括的统计模型。到序列空间中看不见的区域。此外,该文章还坚持认为统计模型“在语义上是丰富的,并且在结构,进化和生物上都有扎实的基础”。
文章的作者写道:“我们的数据驱动方法可以预测天然和从头设计的蛋白质的稳定性,以及分子多样性突变体的定量功能,这与最新方法具有竞争性。” “ UniRep进一步使蛋白质工程任务的效率提高了两个数量级。”
蛋白质工程的更多常规方法包括定向进化和合理设计。在定向进化中,蛋白质工程师随机改变编码天然蛋白质的氨基酸构件的线性序列,并筛选具有所需活性的变体。在合理的设计中,蛋白质工程师根据蛋白质的实际3D结构对蛋白质建模,以识别可能会影响蛋白质功能的氨基酸。
定向进化只能覆盖可能的蛋白质序列巨大空间的一小部分。精心设计的3D蛋白质结构的相对稀缺性限制了合理的设计方法。但是,UniRep承诺对蛋白质功能有更全面的了解。
“无需广泛表征蛋白质来理解其设计原理,我们通过在公共数据库中系统地寻找大量原始蛋白质序列中的模式,而是使用神经网络以无偏见的方式学习那些规则,”研究生Surojit Biswas说是Church小组的学生,也是《自然方法》论文的三位共同第一作者之一。“神经网络通过许多艰苦的研究,学到了许多人类以前知道的规则,除此之外,它还发现了蛋白质的新功能。”
可以将神经网络方法比喻为学习一种语言,在这种语言中,学习者可以建立语义理解,了解如何从字母和单词的字符串构造复杂的句子。在蛋白质语言中,UniRep经过培训,可以探索公共数据库中包含的蛋白质序列中的所有可能性,从而从其第一个氨基酸开始预测蛋白质序列中的下一个氨基酸。
在重复处理蛋白质的其余部分(一次一个氨基酸)的过程中,UniRep制作并利用了迄今为止在蛋白质中看到的序列的内部“摘要”,该小组称其为“隐藏状态”,考虑到其个体顺序和结构特征。将这些信息以及来自许多其他蛋白质的结果反馈回其算法,UniRep逐渐修改了其构造隐藏状态的方式,从而随着时间的推移提高了其预测能力。
在语言类比中,基于对语法和单词选择的不断改进,学习者将能够以更高的可能性预测他们正在阅读的句子的下一个单词。
“我们在大约三周的时间内对UniRep进行了约2400万个蛋白质序列的培训,以使其能够预测序列及其与诸如蛋白质稳定性,二级结构以及内部序列对蛋白质内周围溶剂的可及性之类的特性之间的联系,” Grigory Khimulya是哈佛大学的学生,也是Biswas和Ethan C. Alley的共同第一作者。“ UniRep准确地描述了来自非常不同的蛋白质家族的蛋白质的这些特征,这些蛋白质的结构在先前的研究中得到了很好的表征,甚至在本质上没有对应物的合成蛋白质中也是如此。”
该团队将UniRep更进一步,并将其用作预测单个氨基酸取代如何影响蛋白质功能的工具。想想疯子,但对于蛋白质。
该神经网络以多种生物学功能(包括酶催化,DNA结合,分子传感)可靠地量化了8种不同蛋白质中单个氨基酸突变的影响。此外,他们使用维多利亚水母绿色荧光蛋白(GFP)作为模型,委托UniRep分析该蛋白的64,800个变异体,每个变异体带有1–12个突变,这表明它可以准确预测突变的分布和相对负担改变了蛋白质的亮度。
丘奇说:“与其他策略相比,我们的数据驱动方法在预测蛋白质的多种特性方面达到了最新或更高的性能,而成本却远低于其他方法。” “这使它成为许多领域蛋白质工程师的真正授权工具。”
推荐内容
-
11月19日江苏疫情最新数据公布 江苏昨日新增境外输入无症状感
江苏11月18日新增境外输入无症状感染者1例。据消息显示,11月18日0-24时,江苏无新增确诊病例,新增境外输入无症状感染者1例。目前,在定点
-
今日四川疫情最新消息 新增境外输入确诊病例1例
四川疫情今日疫情具体详情如何?想必大家都想值得,根据四川省卫健委官方微博消息,昨日四川新增境外输入确诊病例1例,下面我们一起来看...
-
研究人员发现了一种威胁南极洲最丰富的海星的疾病
由巴塞罗那大学生物学院和生物多样性研究所(IRBio)的专家领导的一项研究已经确定了一种影响海星Odontaster validus的疾病,这是南极海底最
-
豆神教育现状如何最新消息还有希望吗?豆神大语文最近什么情况?
【导读】你知道豆神教育吗?据消息显示,豆神教育,全称是豆神教育科技(北京)股份有限公司,是一家在深圳证券交易所上市的公司。13日晚,豆
-
李靓蕾王力宏现状最新消息:李靓蕾遭王力宏死亡威胁事件是怎么回
想必这段时间,大家对于李靓蕾遭王力宏死亡威胁一事是非常关注的。具体是什么情况呢?为什么会发生这样的事呢?下面跟小编一起来了解下。...
-
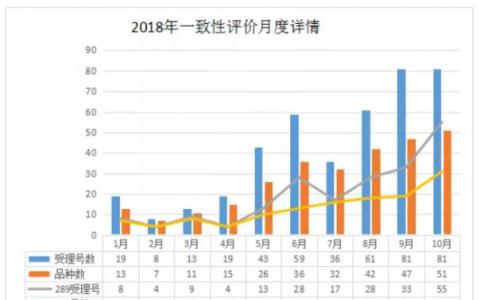
10月一致性评价快速推进阿 阿莫西林胶囊成爆款品种
2018年无疑已经进入倒计时模式,在一致性评价关键性的一年里,CDE每一次关于一致性评价动态的更新都牵动着医药人的心。10月一致性评价更是
-
李佳琦时尚先生封面 年赚2亿是怎么办到的?
【导读】说到李佳琦大家都会知道是淘宝主播,但是也因为优质的外表加上自身的努力让李佳琦在网络爆红,今日李佳琦时尚先生封面公布感兴...
-
广州疫情最新消息现在是什么风险区?广州疫情哪些地方被列为封控区
【摘要】非必要不外出、不出行、不远行。如果您28天内有境外或14天内有国内中高风险区的旅居史,请及时向社区报告。2022广州疫情最新消息截
-
新冠疫苗对感染奥密克戎毒株有用吗?奥密克戎毒株传染性怎么样
【提醒】当前疫情形势严峻复杂,防控不能放松。要时刻保持个人防护意识,支持配合防控措施,主动接种新冠病毒疫苗。11月29日电 综合报道,
-
上海股神杨百万去世的死因是什么?杨百万得了什么病?个人资料简
上海股神杨百万死因揭晓,杨百万究竟得了什么病去世?而对于中国第一股民杨百万去世的这个话题,今天的你是否也在关注着?究竟什么情况?下面